Blog
本記事は、2018年インターンシップ・PEとして勤務した Kaushalya Madhawa さんによる寄稿です。
本記事はこちらの英語ブログの日本語訳です。
本記事では、私たちが最近投稿した論文 “GraphNVP: An Invertible Flow Model for Generating Molecular Graphs” を紹介します。
コードも公開しています → Github repo
分子の生成モデル
望ましい薬理学的な物性値をもつ新しい分子の発見は創薬の分野において重要な問題です。これまで、候補となる化合物を実際に合成して実験を行うといったことがされてきました。
しかし、化合物のとりえる形の可能性は膨大でそれらを合成し、実験するということはとても時間がかかります。
このように望む物性値をもつ分子を探す代わりに、”de novo drug design” では、新しい化合物を興味対象とする物性値とともに設計するということを行います。
近年の深層学習の技術発展、特に深層生成モデルの分野は “de novo drug design” にとって重要な技術となってきています。
分子の表現方法
深層学習で分子の生成モデルを考える際、初めの重要なステップはどのように化合物を表現するかということです。
初期の研究では SMILES という、文字列ベースの表現方法を採用していました。
これらの研究は、 RNN ベースの言語モデルや、Variational AutoEncoder (VAE) を用いることで、SMILES の文字列を生成しそれを分子に変換します。
このアプローチの問題点はSMILES 文字列での小さな変化が分子全体でみると大きく変化するような場合があるということです。
そこで、分子をグラフを用いて表現しようというアプローチが出てきました。この問題は “molecular graph generation” として扱われます。
分子は無向グラフとして表現されます。原子をノード、結合 (bond) を辺に対応させます。この表記を用いると分子の構造・結合情報は Adjacency tensor (隣接テンソル) \( A \) として、また各ノードの原子(酸素、フッ素など)を node feature matrix \( X \) で表すことができます。この定式化の下で、分子の生成問題はグラフの生成問題として扱うことができ、これまでに技術発展してきた GANやVAEといった深層学習のモデルを用いることができます。さらにグラフの生成問題は2つのタイプに分類することができます。1つめは分子のグラフを”順番に” 生成する方法で ノード (原子) や 辺 (結合) を1つずつ生成していきます。もう1つの方法が直接グラフを生成するタイプで、画像の生成と似たように一度に全体を生成します。
可逆性
上記で紹介したVAEやGANと比較して、可逆な Flow を用いたモデルは尤度の最大化を直接行えるという長所があります。
さらに、Flow モデルは可逆な変換のみで構成されているので、逆変換が可能で100% 分子→潜在変数→分子 の再構成を行うことができます。
GANのみの生成モデルなどEncoder がない場合は、特定の分子を操作して生成・最適化を行うことは困難です。例えば、特定のベースとなる分子 (query) に似ている分子を生成して最適化を行っていくということが (創薬の分野で Lead optimization と呼ばれる) 困難です。一方Flow モデルは 分子の潜在変数を直接計算できるので、query 分子に対する潜在変数を簡単に求めることができそこから最適化を行うことも可能です。
提案モデル
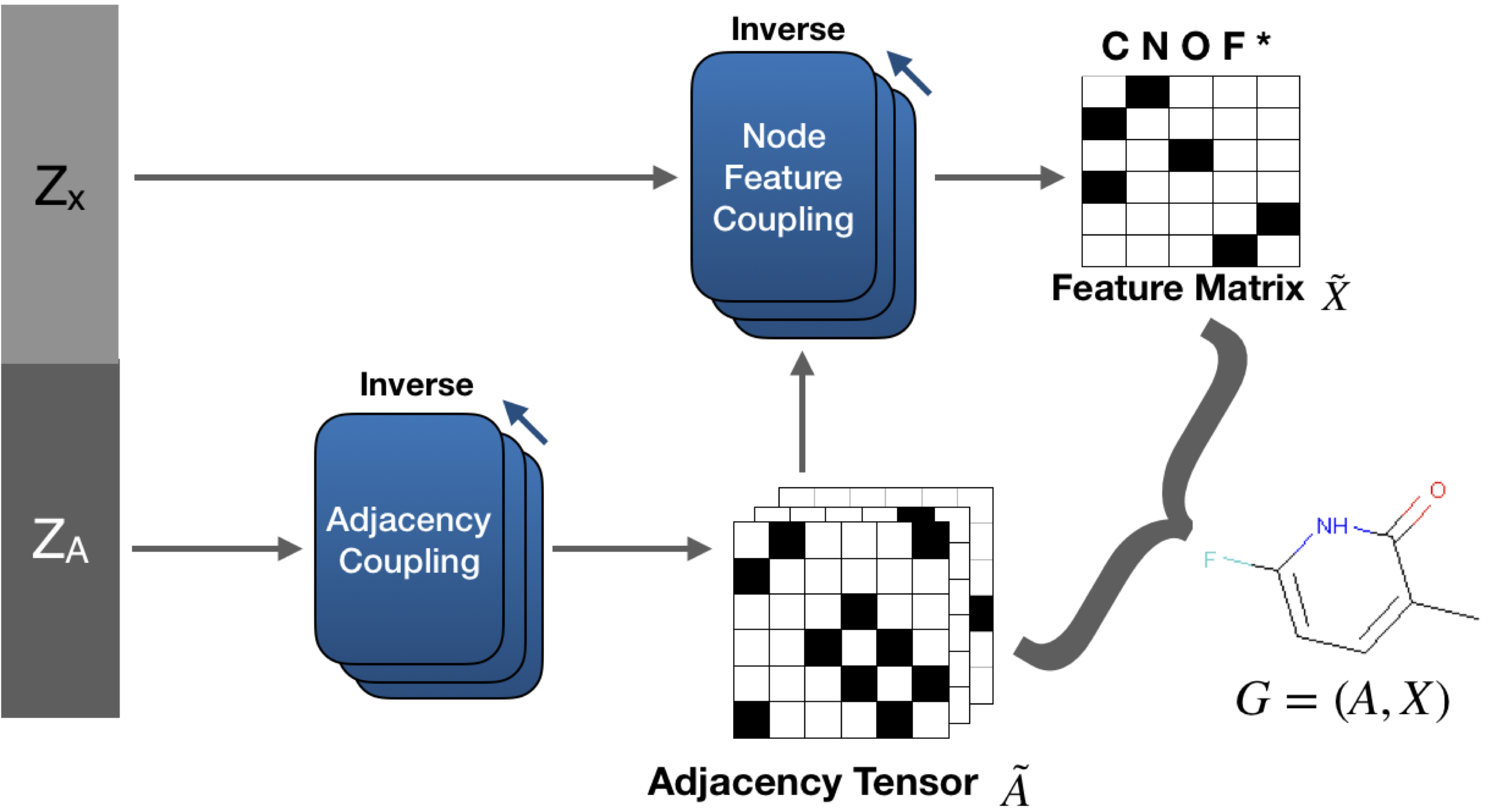
提案モデルであるGraphNVPは上図のような構成をとります。GraphNVPは私たちの知る限りではOne-shot Graph生成モデルとして初めてFlow を用いたモデルです。
このモデルは辺に対応する変数とノードに対応する変数の2つの潜在変数を使用し、グラフ構造や原子種類の分布を表現できるようにします。
これらのをFlowの定式下で計算できるよう、 Adjacency Coupling および Node Feature Coupling と呼ばれる2つのCoupling layerを提案しました。
グラフの生成を行う際は、まずAdjacency tensorを生成し、次に Node feature tensor を graph convolutional network を用いて生成します。
結果
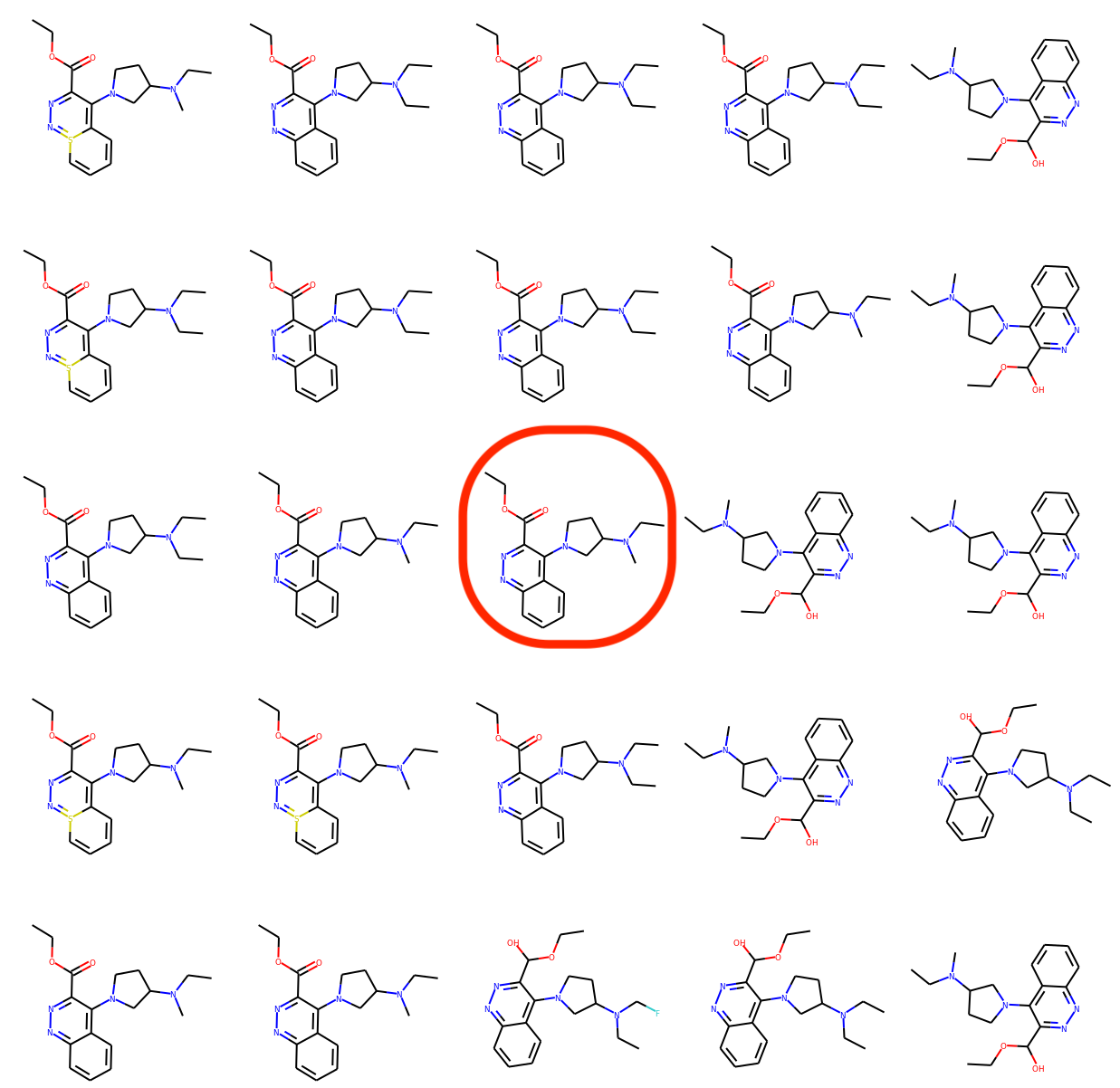
訓練データから分子をとり、その潜在変数 \( z_0 \) をGraphNVPで計算します。そして潜在空間上でランダムに選んだ2つの方向の軸に対して \( z_0\) が中心となるような2次元グリッドを構成し、その各点で潜在変数をデコードして分子を生成したものが下図となります。
下図の可視化結果では、似たような分子が並んでおり、隣どおしでは少しの原子が変わっているだけというようなものとなっており、提案モデルがスムーズな潜在空間の学習をできていることが見て取れます。
さいごに:メンターより
Kaushalyaさんのメンターを担当した、中郷・石黒です。
今回の研究は2018年夏のインターンシップから始めたものです。このころはGraphの生成モデルの研究が盛んになり様々なアプローチでの生成モデルが提案され始めたころでしたが、まだFlowを用いたグラフの生成モデルはないので取り組んでみたいというKaushalyaさんの持ち込みからはじまったものでした。
グラフ生成モデルとしては初めての取り組みであり、またFlowを用いるモデルはかなり深いネットワークを使用する必要があり計算量も大きくなるということで、研究には時間がかかりましたが、最終的には論文として形にすることができ、コードも公開することができました。
最後に宣伝となりますが、PFNでは今回の “Drug Discovery / Material Discovery” の分野だけでなく、多種多様な業界で機械学習適用の横断プロジェクトを実施しており、中途・新卒の人材募集も通年で行っております!
興味のある方はぜひこちらのページをご覧ください。